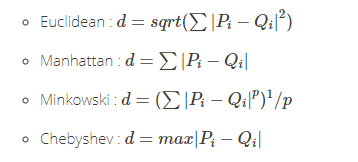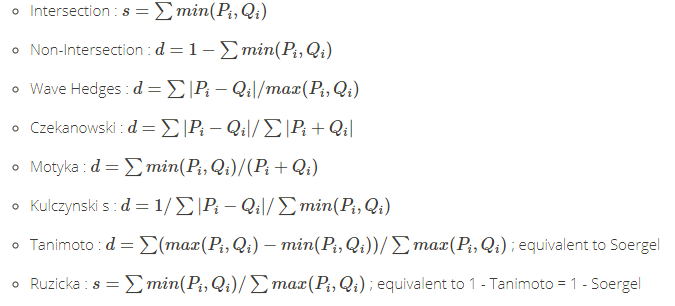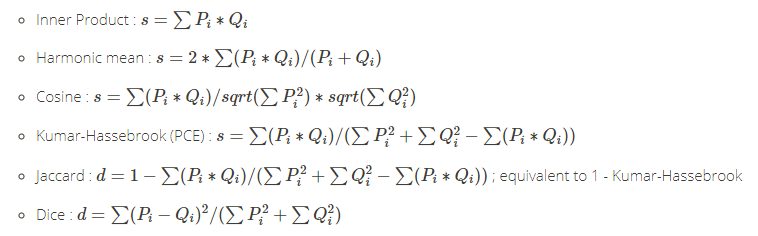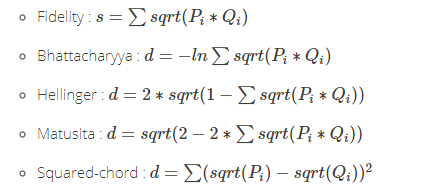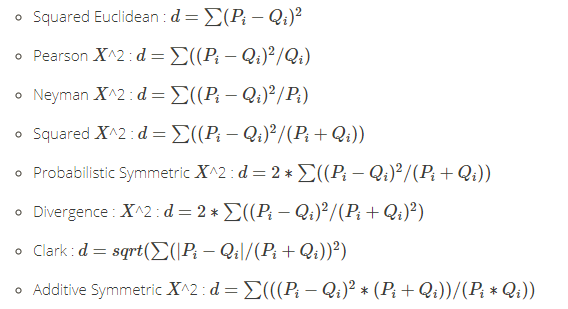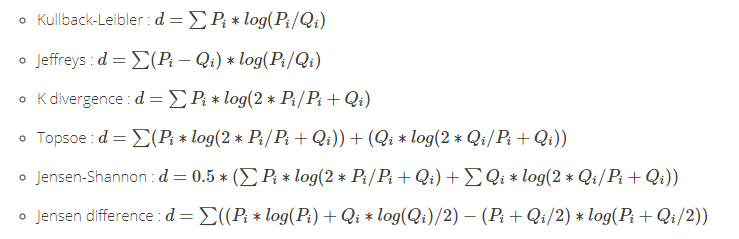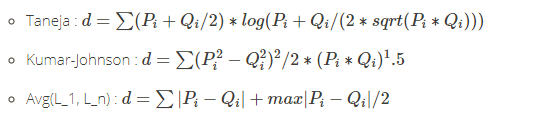R的极客理想系列文章,涵盖了R的思想,使用,工具,创新等的一系列要点,以我个人的学习和体验去诠释R的强大。
R语言作为统计学一门语言,一直在小众领域闪耀着光芒。直到大数据的爆发,R语言变成了一门炙手可热的数据分析的利器。随着越来越多的工程背景的人的加入,R语言的社区在迅速扩大成长。现在已不仅仅是统计领域,教育,银行,电商,互联网….都在使用R语言。
要成为有理想的极客,我们不能停留在语法上,要掌握牢固的数学,概率,统计知识,同时还要有创新精神,把R语言发挥到各个领域。让我们一起动起来吧,开始R的极客理想。
关于作者:
- 张丹(Conan), 程序员/Quant: Java,R,Nodejs
- blog: http://blog.fens.me
- email: bsspirit@gmail.com
转载请注明出处:
http://blog.fens.me/r-distance
前言
距离算法是做数据挖掘常用的一类算法,距离算法有很多种,比如欧式距离、马氏距离、皮尔逊距离,距离算法主要应用在计算数据集之间关系。本文用R语言来philentropy包,实现多种距离的算法,很多可能是大家完全没有听过的,让我们在开拓一下知识领域吧。
目录
- 距离算法包philentropy
- 46种距离算法详解
- 距离函数的使用
1.距离算法包philentropy
在做距离算法调研时,无意中发了philentropy包。它实现了46个不同距离算法和相似性度量,通过不同数据的相似度比较,为基础研究提供了科学基础。philentropy包,为聚类、分类、统计推断、拟合优度、非参数统计、信息理论和机器学习提供了核心的计算框架,支持基于单变量或者多变量的概率函数的计算。
philentropy包主要包括了2种度量的计算方法,距离度量和信息度量。本文介绍距离度量的使用,对于信息度量的使用,请参考文章R语言实现信息度量。
philentropy项目github地址:https://github.com/HajkD/philentropy
本文的系统环境为:
- Win10 64bit
- R: 3.4.2 x86_64-w64-mingw32
安装philentropy包,非常简单,一条命令就可以了。
~ R
> install.packages("philentropy")
> library(philentropy)
查看距离算法列表
> getDistMethods()
[1] "euclidean" "manhattan" "minkowski" "chebyshev"
[5] "sorensen" "gower" "soergel" "kulczynski_d"
[9] "canberra" "lorentzian" "intersection" "non-intersection"
[13] "wavehedges" "czekanowski" "motyka" "kulczynski_s"
[17] "tanimoto" "ruzicka" "inner_product" "harmonic_mean"
[21] "cosine" "hassebrook" "jaccard" "dice"
[25] "fidelity" "bhattacharyya" "hellinger" "matusita"
[29] "squared_chord" "squared_euclidean" "pearson" "neyman"
[33] "squared_chi" "prob_symm" "divergence" "clark"
[37] "additive_symm" "kullback-leibler" "jeffreys" "k_divergence"
[41] "topsoe" "jensen-shannon" "jensen_difference" "taneja"
[45] "kumar-johnson" "avg"
46个距离算法,有一些是我们常用的比如:euclidean,manhattan,minkowski,pearson, cosine,squared_chi, 其他的我也不知道,正好拓宽知识,好好学习一下。
philentropy包的函数,其实很简单,只有14个,大量的算法其实都已经被封装到distance()函数中,直接使用distance()函数就行完成各种算法的计算,让我们使用起来会非常方便。我们来看一下,函数列表:
- distance(): 计算距离
- getDistMethods(),获得距离算法列表
- dist.diversity(),概率密度函数之间的距离差异
- estimate.probability(),从计数向量估计概率向量
- lin.cor(),线性相关性判断
- H(): 香农熵, Shannon’s Entropy H(X)
- JE() : 联合熵, Joint-Entropy H(X,Y)
- CE() : 条件熵, Conditional-Entropy H(X|Y)
- MI() : 互信息, Shannon’s Mutual Information I(X,Y)
- KL() : KL散度, Kullback–Leibler Divergence
- JSD() : JS散度,Jensen-Shannon Divergence
- gJSD() : 通用JS散度,Generalized Jensen-Shannon Divergence
- binned.kernel.est(),实现了KernSmooth包提供的核密度估计函数的接口
从函数列表来看,主要分为3种类别的函数,第一类是距离测量的函数,包括distance(),
getDistMethods(), dist.diversity(), lin.cor()和 estimate.probability()。第二类是相关性分析,包括lin.cor()函数。第三类是信息度量函数H(),JE(),CE(),MI(),KL(),JSD(),gJSD()。信息度量函数的使用,请参考文章R语言实现信息度量。
2. 46种算法详解
接下来,就让我们深入每个算法吧,从名字到公式,再到函数使用,最后到使用场景。
距离算法列表:
- euclidean:欧式距离,是一个通常采用的距离定义,在m维空间中两个点之间的真实距离,或者向量的自然长度(即该点到原点的距离)。
- manhattan:曼哈顿距离,用于几何空间度量,表示两个点在标准坐标系上的绝对轴距距离总和。
- minkowski:闵可夫斯基距离,是欧氏空间中的广义距离函数,其参数p值的不同代表着对空间不同的度量。
- chebyshev:切比雪夫距离,是向量空间中的一种度量,二个点之间的距离定义是其各坐标数值差绝对值的最大值。
- sorensen:测量每个样本单位,对单位总数的距离测量贡献度,广告用于生态学。
- gower:高尔距离,将向量空间缩放为规范化空间,可计算逻辑值,数字,文本的距离,距离结果为0到1之间的数字。
- soergel:测量每个样本单位,对最大值总数的距离测量贡献度。
- kulczynski:与soergel相反,测量每个样本单位,对最小值总数的距离测量贡献度。
- canberra:堪培拉距离,是矢量空间中的点对之间的距离的数值度量,它是L_1距离的加权版本。
- lorentzian:洛伦兹距离,绝对的差异并应用自然对数。
- intersection:交叉距离,最小轨道交叉距离,是天文学中用于评估天文物体之间潜在的近距离接近和碰撞风险的度量。它被定义为两个物体的密切轨道的最近点之间的距离。
- non-intersection:非交叉距离
- wavehedges:波浪距离,
- czekanowski:
- motyka:莫蒂卡方程,是czekanowski的一半。
- kulczynski_s:
- tanimoto:是标准化内积的另一种变体。
- ruzicka:
- inner_product:内部产品空间,计算两个向量的内积产生标量,有时称为标量积或点积。
- harmonic_mean:调和平均值。
- cosine:余弦距离,是用向量空间中两个向量夹角的余弦值作为衡量两个个体间差异的大小的度量。
- hassebrook(PCE):利用P•Q来测量能量的峰值,简称PCE。
- jaccard:杰卡德距离,用于计算样本间的相似度,分子是A和B的交集大小,分母是A和B的并集大小。
- dice:骰子
- fidelity:保真度,在量子信息理论中,保真度是两个量子态“接近”的度量。
- bhattacharyya:巴氏距离,测量两个概率分布的相似性。
- hellinger:海林格,用来度量两个概率分布的相似度,它是F散度的一种。
- matusita:
- squared_chord:
- squared_euclidean:欧式距离的平方
- pearson:皮尔森距离,分子是两个集合的交集大小,分母是两个集合大小的几何平均值,是余弦距离的一种变型。
- neyman:奈曼,
- squared_chi:
- prob_symm:
- divergence:散度,
- clark:克拉克,
- additive_symm:算术和几何平均散度.
- kullback-leibler:KL散度,用于计算相熵或信息偏差,是衡量两个分布(P、Q)之间的距离,越小越相似。
- jeffreys:杰弗里斯,J分歧。
- k_divergence:K散度,
- topsoe:托普索,是k_divergence加法的对称形式。
- jensen-shannon:詹森香农,是topsoe距离的一半。
- jensen_difference:
- taneja:塔内加,计算算术和几何平均偏差。
- kumar-johnson:库马尔-约翰逊,
- avg:平均
由于精力和基础知识有限,对每一种算法还没有更深入的理解和使用,后面会继续补充。这些距离的详细解释,请参考文章 http://csis.pace.edu/ctappert/dps/d861-12/session4-p2.pdf
这么多种的距离算法,其实可以分成8大距离家族,每个家族中不同的算法思路是类似的,可以通过变形或参数不同赋值,进行算法的相互转换。
L_p Minkowski家族,通过对Minkowski 算法p值的不同赋值,可以转换成不同的算法,当p=1时Minkowski距离转为曼哈顿距离;当p=2变Minkowski距离转为欧氏距离;当p接近极限最大值时,Minkowski距离是转为切比雪夫距离。
L_1家族,用于准确的测量绝对差异的特征。
Intersection 交叉距离家族,用于交叉点之间的相似度变换。
Inter Product 家族,几何空间的相似性度量,用于特定的 P•Q 变量来计算。
Squared-chord 家族,在量子信息理论中,量子态“接近”的度量。
Squared L_2 家族( X^2 Squared 家族),以平方的欧几里得距离做为被除数。
香浓信息熵家族,信息熵偏差测量。
组合公式,利用多种算法思路,进行组合的距离测量方法。
3. 距离函数的使用
了解了这么多的距离算法后,让我们来使用一下philentropy包强大的功能函数,把算法落地。
3.1 distance()函数使用
distance()函数,用来计算两个概率密度函数之间的距离和相似度,上面所列出的所有的距离算法都被封装在了这个函数里。
distance()函数定义:
distance(x, method = "euclidean", p = NULL, test.na = TRUE, unit = "log", est.prob = NULL)
参数列表:
- x, 数值类型的向量或数据集
- method, 算法的名称
- p, minkowski闵可夫斯基距离的p值,p=1为曼哈顿距离,p=2为欧氏距离,p取极限时是切比雪夫距离
- test.na, 检测数据集是否有NA值,不检测为FALSE,计算会快。
- unit,对数化的单位,依赖于日志计算的距离
- est.prob 从计数估计概率,默认值为NULL
计算euclidean距离,用iris的数据集。
> library(magrittr)
# 查看iris数据集
> head(iris)
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
计算第1个点(第1行)和第2个点(第2行)的euclidean距离,分别使用philentropy包的distance(),Stats包的dist(),和自己通过公式计算。
# 使用distance()函数
> dat1<-iris[1:2,-5]
> distance(dat1, method="euclidean")
Metric: 'euclidean' using unit: 'log'.
euclidean
0.5385165
# 再使用系统自带的dist()函数
> dist(dat1)
1
2 0.5385165
# 公式计算
> (dat1[1,]-dat1[2,])^2 %>% sum %>% sqrt
[1] 0.5385165
3种方法,计算的结果是完全一致。
接下来,我们构建一个iris的距离矩阵,为了展示清楚,我们选iris的前6个点来计算距离。分别使用distance()和dist()函数。
> dat2<-head(iris[,-5])
# 距离矩阵
> distance(dat2)
Metric: 'euclidean' using unit: 'log'.
v1 v2 v3 v4 v5 v6
v1 0.0000000 0.5385165 0.509902 0.6480741 0.1414214 0.6164414
v2 0.5385165 0.0000000 0.300000 0.3316625 0.6082763 1.0908712
v3 0.5099020 0.3000000 0.000000 0.2449490 0.5099020 1.0862780
v4 0.6480741 0.3316625 0.244949 0.0000000 0.6480741 1.1661904
v5 0.1414214 0.6082763 0.509902 0.6480741 0.0000000 0.6164414
v6 0.6164414 1.0908712 1.086278 1.1661904 0.6164414 0.0000000
# 下三角距离矩阵
> dist(dat2)
1 2 3 4 5
2 0.5385165
3 0.5099020 0.3000000
4 0.6480741 0.3316625 0.2449490
5 0.1414214 0.6082763 0.5099020 0.6480741
6 0.6164414 1.0908712 1.0862780 1.1661904 0.6164414
验证后,我们就可以放心使用distance()函数。通过对比实验,我们可以很快的学习并使用各种距离算法。
3.2 dist.diversity()函数
dist.diversity()函数,用来计算所有距离的值。由于有一些距离有对于数据集本身的要求,所以我们需要构建一个能适应所有距离算法的数据集。
# 生成数据集,2个点,10个维度
> P <- 1:10/sum(1:10)
> Q <- 20:29/sum(20:29)
> x <- rbind(P,Q)
# 打印数据集
> head(x)
[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8]
P 0.01818182 0.03636364 0.05454545 0.07272727 0.09090909 0.1090909 0.1272727 0.1454545
Q 0.08163265 0.08571429 0.08979592 0.09387755 0.09795918 0.1020408 0.1061224 0.1102041
[,9] [,10]
P 0.1636364 0.1818182
Q 0.1142857 0.1183673
使用dist.diversity()函数计算所有的距离。
> dist.diversity(x,p=2)
euclidean manhattan minkowski chebyshev sorensen
0.12807130 0.35250464 0.12807130 0.06345083 0.17625232
gower soergel kulczynski_d canberra lorentzian
0.03525046 0.29968454 0.42792793 2.09927095 0.34457827
intersection non-intersection wavehedges czekanowski motyka
0.82374768 0.17625232 3.16657887 0.17625232 0.58812616
kulczynski_s tanimoto ruzicka inner_product harmonic_mean
2.33684211 0.29968454 0.70031546 0.10612245 0.94948528
cosine hassebrook jaccard dice fidelity
0.93427641 0.86613103 0.13386897 0.07173611 0.97312397
bhattacharyya hellinger matusita squared_chord squared_euclidean
0.02724379 0.32787819 0.23184489 0.05375205 0.01640226
pearson neyman squared_chi prob_symm divergence
0.16814418 0.36742465 0.10102943 0.20205886 1.49843905
clark additive_symm kullback-leibler jeffreys k_divergence
0.86557468 0.53556883 0.09652967 0.22015096 0.02922498
topsoe jensen-shannon jensen_difference taneja kumar-johnson
0.05257867 0.02628933 0.02628933 0.02874841 0.62779644
avg
0.20797774
3.3 estimate.probability()函数
estimate.probability()函数,采用数字计数来计算向量的估计概率。estimate.probability()函数方法实现,目前只有一个方法实现,计算每个值占合计的比率,其实就是一种数据标准归的计算方法。
> estimate.probability
function (x, method = "empirical")
{
if (!is.element(method, c("empirical")))
stop("Please choose a valid probability estimation method.")
if (method == "empirical") {
return(x/sum(x))
}
}
>environment: namespace:philentropy<
我们新建一个向量,用estimate.probability()函数,来计算向量的估计概率。
# 新建x1向量
> x1<-runif(100);head(x1)
[1] 0.6598775 0.2588441 0.5329965 0.5294842 0.8331355 0.3326702
# 计算估计概率
> x2<-estimate.probability(x1);head(x2)
[1] 0.013828675 0.005424447 0.011169702 0.011096097 0.017459543 0.006971580
# 打印统计概率
> summary(x2)
Min. 1st Qu. Median Mean 3rd Qu. Max.
0.0002181 0.0044882 0.0096318 0.0100000 0.0153569 0.0206782
# 画散点图
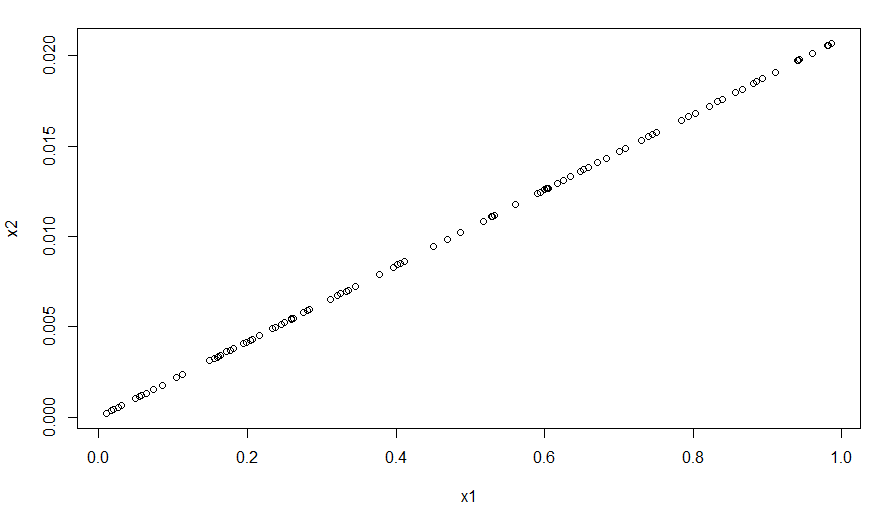
> plot(x1,x2)
从图中看到,整个数据分布在对角线上,x1为均匀分布生成的向量,x2为x1的估计概率,仅仅做了做数据值域进行了缩放,并没有影响数据的分布变化和线性特征,其实就是对数据做了一个标准化的过程。
3.4 lin.cor()函数
lin.cor()函数,用来计算两个向量之间的线性相关,或计算矩阵的相关矩阵。
函数定义:
lin.cor(x, y = NULL, method = "pearson", test.na = FALSE)
参数列表:
- x,变量1
- y,变量2,与x进行比较
- method,相关性算法的名称,默认为pearson距离算法,支持5种算法分为是pearson,pearson2,sq_pearson,kendall,spearman
- test.na, 检测数据集是否有NA值,不检测为FALSE,计算会快。
相关性计算,最大值1为完全正相关,最小值-1为完全负相关,0为不相关。我们来创建数据集,进行相关性的测试。
# 创建向量x1,x2,x3
> x1<-runif(100)
> x2<-estimate.probability(x1)
> x3<-rnorm(100)
# 判断x1,x2的相关性,pearson 皮尔森相关系数
> lin.cor(x1,x2)
pearson
1
# 判断x1,x3的相关性,pearson 皮尔森相关系数
> lin.cor(x1,x3)
pearson
0.0852527
# 判断x1,x3的相关性,pearson2 皮尔森非集中的相关系数
> lin.cor(x1,x3,method = 'pearson2')
pearson2
0.01537887
# 判断x1,x3的相关性,sq_pearson 皮尔森平方的相关系数
> lin.cor(x1,x3,method = 'sq_pearson')
sq_pearson
0.00151915
# 判断x1,x3的相关性,kendall 肯德尔相关系数
> lin.cor(x1,x3,method = 'kendall')
kendall
0
# 判断x1,x3的相关性,spearman 斯皮尔曼相关系数
> lin.cor(x1,x3,method = 'spearman')
spearman
0
通过lin.cor()函数,可以快速进行线性相关性的验证,非常方便。
本文重点介绍了philentropy包,对于距离算法的定义和距离测量的函数的使用。很多的距离算法我也是第一次学习,知识需要积累和总结,本文不完善的内容,后面我们找时间再进行补充。如果本文描述有不当的地方,也请各位朋友,给予指点,让我们一起把知识进行积累。
下一篇文章我们将介绍信息度量函数的使用和算法,请大家继续阅读文章用R语言实现信息度量。
转载请注明出处:
http://blog.fens.me/r-distance