R的极客理想系列文章,涵盖了R的思想,使用,工具,创新等的一系列要点,以我个人的学习和体验去诠释R的强大。
R语言作为统计学一门语言,一直在小众领域闪耀着光芒。直到大数据的爆发,R语言变成了一门炙手可热的数据分析的利器。随着越来越多的工程背景的人的加入,R语言的社区在迅速扩大成长。现在已不仅仅是统计领域,教育,银行,电商,互联网….都在使用R语言。
要成为有理想的极客,我们不能停留在语法上,要掌握牢固的数学,概率,统计知识,同时还要有创新精神,把R语言发挥到各个领域。让我们一起动起来吧,开始R的极客理想。
关于作者:
- 张丹(Conan), 程序员Java,R,Nodejs
- weibo:@Conan_Z
- blog: http://blog.fens.me
- email: bsspirit@gmail.com
转载请注明出处:
http://blog.fens.me/r-install-offline
前言
最近经常去有网络隔离的机构,每次安装R语言的环境都是特别痛苦的经历。物理网络完全隔绝,不能通过互联网进行R语言的安装,只能预先下载所有软件,再进行安装。
对于有众多依赖的库,安装过程就变得很复杂了。无意中,发现了一个可以找到每个包的依赖包的库miniCRAN,让这个安装包过程,变得自动化一点。
目录
- 离线安装R的依赖库的过程
- miniCRAN介绍
- 5分钟上手
- miniCRAN包的函数介绍
- miniCRAN的使用方法
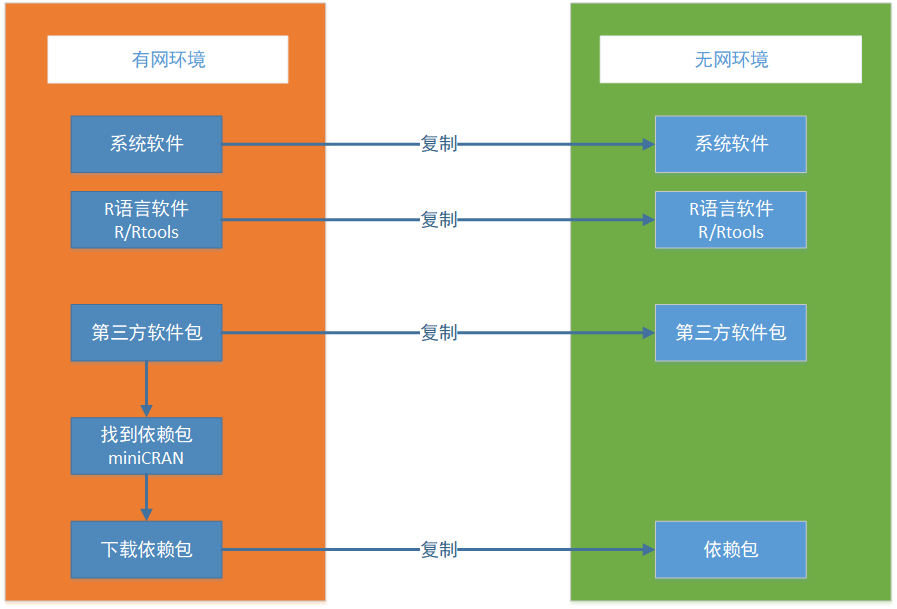
1. 离线安装R的依赖库的过程
对于物理网络完全被隔绝的情况,我们不能通过互联网进行R语言的安装,只能预先下载所有软件,再进行安装。
操作过程包括以下几步操作:
- 在有网的环境中,下载系统软件,比例 windows环境要下载对应的.net framework。
- 在有网的环境中,下载R语言软件。
- 在有网的环境中,下载第三方软件包,比如xts包。
- 在有网的环境中,用miniCRAN找到xts包,所有的依赖包。
- 在有网的环境中,下载所有的依赖包。
- 通过U盘或者其他的方式,复制到无网的环境中。
- 分别安装系统软件,R语言软件,依赖包,软件包。
- 重复以上过程,直到把软件都安装好。
以上操作过程中,最复杂的操作就在于找到所有的依赖包。
2. miniCRAN介绍
miniCRAN包,主要用于发现软件包的依赖关系。R语言中,每个包一般都其所依赖的扩展包,对于企业或者机构内网来说,由于防火墙或者基于安全的管理规则,不允许从互联网下载安装。那么,用户就需要从CRAN中,下载对应的软件包,一个一个安装。通过miniCRAN包,可以递归读取这些软件包的依赖关系,然后就可以下载这些依赖包了。
开发环境所使用的系统环境
- Win10 64bit
- R: 3.2.3 x86_64-w64-mingw32/x64 b4bit
miniCRAN包的安装比较简单,直接用install.pacakges()函数就行。
> install.packages("miniCRAN")
binary source needs_compilation
miniCRAN 0.2.7 0.2.11 FALSE
installing the source package 'miniCRAN'
URLhttp://mirrors.tuna.tsinghua.edu.cn/CRAN/src/contrib/miniCRAN_0.2.11.tar.gz'
Content type 'application/octet-stream' length 4114313 bytes (3.9 MB)
downloaded 3.9 MB
* installing *source* package 'miniCRAN' ...
** 'miniCRAN'MD5
** R
** data
*** moving datasets to lazyload DB
** inst
** preparing package for lazy loading
** help
*** installing help indices
** building package indices
** installing vignettes
** testing if installed package can be loaded
*** arch - i386
*** arch - x64
* DONE (miniCRAN)
3. 5分钟上手
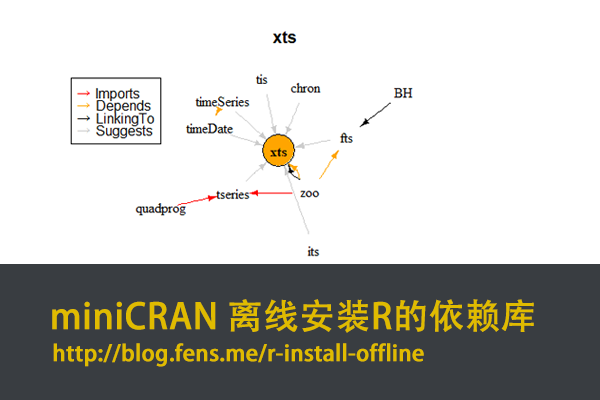
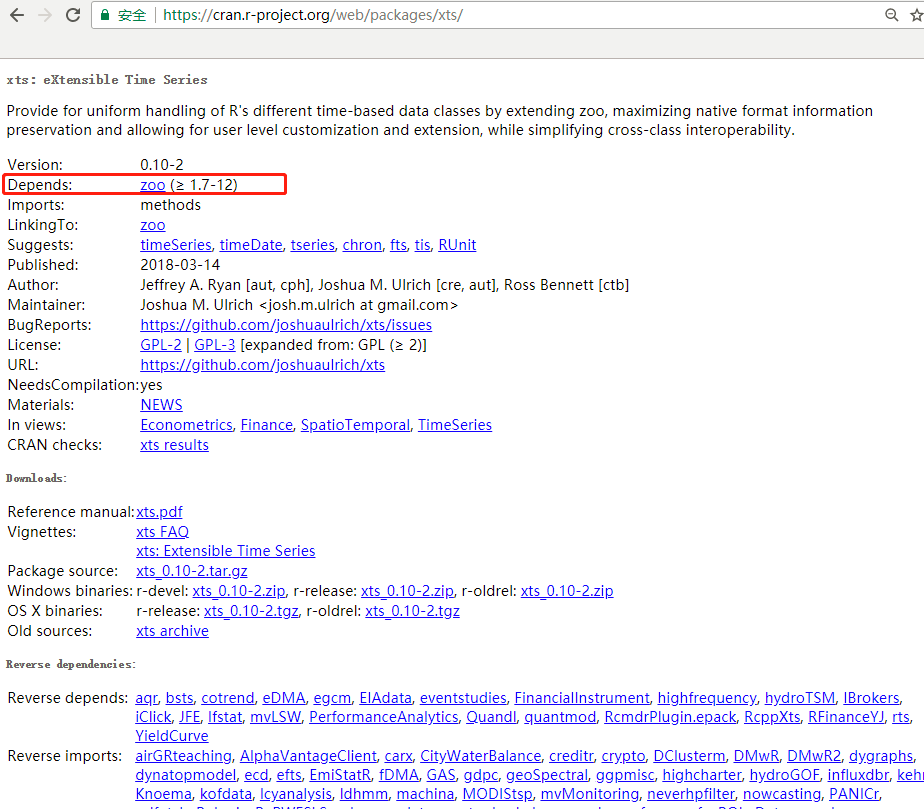
我们先做一个小的测试,分析一下chron包如果本地安装,都需要使用哪些包,会有哪些依赖关系。我们打开CRAN上面的,官方项目网页,https://cran.r-project.org/web/packages/xts/。
xts唯一强依赖的包(depends)是zoo,引用R系统包methods,同时建议安装timeSeries, timeDate, tSeries, chron, fts, tis, RUnit包。
接下来,我们通过miniCRAN找到xts包的依赖关系。
> library(miniCRAN)
> tags <- "xts"
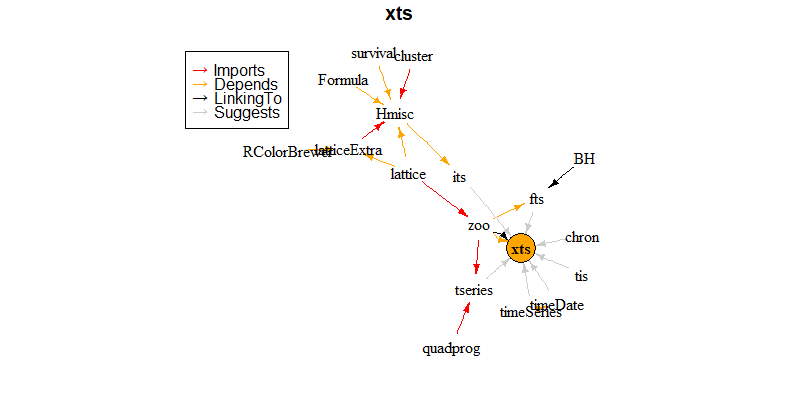
> pkgDep(tags, availPkgs = cranJuly2014)
[1] "xts" "zoo" "lattice"
[4] "timeDate" "quadprog" "Hmisc"
[7] "survival" "Formula" "latticeExtra"
[10] "cluster" "RColorBrewer" "BH"
[13] "timeSeries" "tseries" "its"
[16] "chron" "fts" "tis"
画出依赖关系图。
> dg <- makeDepGraph(tags, enhances = TRUE, availPkgs = cranJuly2014)
> plot(dg, legendPosition = c(-1, 1), vertex.size = 20)
从图中,可以把整个的依赖关系看得很清楚了。
4. miniCRAN包的函数介绍
整个miniCRAN包大概有20多个函数,其中最有用的函数就是pkgDeg()和pkgAvail()。
4.1 pkgDeg()函数
pkgDeg()函数,主要对Depends,Imports和LinkLibrary执行递归检索,对Suggests执行非递归检索。
pkgDep(pkg, availPkgs, repos = getOption("repos"), type = "source",
depends = TRUE, suggests = TRUE, enhances = FALSE,
includeBasePkgs = FALSE, Rversion = R.version, quiet = FALSE, ...)
参数解读:
- pkg: 包名
- availPkgs: 所有包列表
- repos: CRAN软件源
- type: 下载类型,源代或二进制
- depends: 依赖关系,包括Depends, Imports, LinkingTo
- suggests: 建议关系
- enhances: 增强关系
- includeBasePkgs: 是否包括R base包
- Rversion: R版本
4.2 pkgAvail()函数
pkgAvail()函数,是对 available.packages()函数的一个封装。如果提供了参数路径,那么该函数将尝试从本地存储库读取数据,否则尝试从repos url的CRAN镜像读取数据。
pkgAvail(repos = getOption("repos"), type = "source",
Rversion = R.version, quiet = FALSE)
参数解读:
- repos: CRAN软件源
- type: 下载类型,源代或二进制
- Rversion: R版本
这个函数在我目前测试的版本中,有一些bug,可以用available.packages()函数来解决。
5. miniCRAN的使用方法
接下来,我们详细地介绍一下,找到某包的依赖的使用方法。
5.1 定义软件源
如果我们用install.packages()函数安装过软件包,在你的本地配置中,会有当前的软件源,那么我们可以用getOption()函数,来获得本地的软件源。
> repos<-getOption("repos")
> repos
CRAN
"https://mirrors.tuna.tsinghua.edu.cn/CRAN/"
CRANextra
"http://www.stats.ox.ac.uk/pub/RWin"
attr(,"RStudio")
[1] TRUE
我当前环境中配置的CRAN软件源为 https://mirrors.tuna.tsinghua.edu.cn/CRAN/ 。
5.2 获得软件源的包列表
通过软件源URL,我们需要获得软件源的包列表。
> curl<-contrib.url(repos)
> aps<-available.packages(curl)
> head(aps)
Package Version Priority Depends Imports LinkingTo
A3 "A3" "1.0.0" NA "R (>= 2.15.0), xtable, pbapply" NA NA
abbyyR "abbyyR" "0.5.1" NA "R (>= 3.2.0)" "httr, XML, curl, readr, plyr, progress" NA
abc "abc" "2.1" NA "R (>= 2.10), abc.data, nnet, quantreg, MASS, locfit" NA NA
ABCanalysis "ABCanalysis" "1.2.1" NA "R (>= 2.10)" "plotrix" NA
abc.data "abc.data" "1.0" NA "R (>= 2.10)" NA NA
abcdeFBA "abcdeFBA" "0.4" NA "Rglpk,rgl,corrplot,lattice,R (>= 2.10)" NA NA
Suggests Enhances License License_is_FOSS License_restricts_use OS_type Archs MD5sum
A3 "randomForest, e1071" NA "GPL (>= 2)" NA NA NA NA NA
abbyyR "testthat, rmarkdown, knitr (>= 1.11)" NA "MIT + file LICENSE" NA NA NA NA NA
abc NA NA "GPL (>= 3)" NA NA NA NA NA
ABCanalysis NA NA "GPL-3" NA NA NA NA NA
abc.data NA NA "GPL (>= 3)" NA NA NA NA NA
abcdeFBA "LIM,sybil" NA "GPL-2" NA NA NA NA NA
NeedsCompilation File Repository
A3 "no" NA "https://mirrors.tuna.tsinghua.edu.cn/CRAN/src/contrib"
abbyyR "no" NA "https://mirrors.tuna.tsinghua.edu.cn/CRAN/src/contrib"
abc "no" NA "https://mirrors.tuna.tsinghua.edu.cn/CRAN/src/contrib"
ABCanalysis "no" NA "https://mirrors.tuna.tsinghua.edu.cn/CRAN/src/contrib"
abc.data "no" NA "https://mirrors.tuna.tsinghua.edu.cn/CRAN/src/contrib"
abcdeFBA "no" NA "https://mirrors.tuna.tsinghua.edu.cn/CRAN/src/contrib"
取出xts包所对应的配置信息。
> aps[which(row.names(aps)=='xts'),]
Package Version
"xts" "0.10-2"
Priority Depends
NA "zoo (>= 1.7-12)"
Imports LinkingTo
"methods" "zoo"
Suggests Enhances
"timeSeries, timeDate, tseries, chron, fts, tis, RUnit" NA
License License_is_FOSS
"GPL (>= 2)" NA
License_restricts_use OS_type
NA NA
Archs MD5sum
NA NA
NeedsCompilation File
"yes" NA
Repository
"https://mirrors.tuna.tsinghua.edu.cn/CRAN/src/contrib"
5.3 找到依赖包的列表
从所有的软件包中,找到对应软件依赖包的列表。
# 要找到xts和TTR软件的依赖包
> libs<-c("xts","TTR")
> pkgList<-pkgDep(pkg=libs,availPkgs=aps,repos=repos)
> pkgList
[1] "xts" "TTR" "zoo" "lattice" "curl" "timeDate" "quadprog"
[8] "quantmod" "BH" "timeSeries" "tseries" "chron" "fts" "tis"
[15] "RUnit"
5.4 下载所有依赖包
运行下载函数,下载所有的依赖包。
> dp<-download.packages(pkgList,"D:/workspace/R/miniCRAN/pgk",type=getOption("pkgType"))
> dp
[,1] [,2]
[1,] "xts" "D:/workspace/R/miniCRAN/pgk/xts_0.10-2.tar.gz"
[2,] "TTR" "D:/workspace/R/miniCRAN/pgk/TTR_0.23-3.tar.gz"
[3,] "zoo" "D:/workspace/R/miniCRAN/pgk/zoo_1.8-1.tar.gz"
[4,] "lattice" "D:/workspace/R/miniCRAN/pgk/lattice_0.20-35.tar.gz"
[5,] "curl" "D:/workspace/R/miniCRAN/pgk/curl_3.2.tar.gz"
[6,] "timeDate" "D:/workspace/R/miniCRAN/pgk/timeDate_3043.102.tar.gz"
[7,] "quadprog" "D:/workspace/R/miniCRAN/pgk/quadprog_1.5-5.tar.gz"
[8,] "quantmod" "D:/workspace/R/miniCRAN/pgk/quantmod_0.4-13.tar.gz"
[9,] "BH" "D:/workspace/R/miniCRAN/pgk/BH_1.66.0-1.tar.gz"
[10,] "timeSeries" "D:/workspace/R/miniCRAN/pgk/timeSeries_3042.102.tar.gz"
[11,] "tseries" "D:/workspace/R/miniCRAN/pgk/tseries_0.10-44.tar.gz"
[12,] "chron" "D:/workspace/R/miniCRAN/pgk/chron_2.3-52.tar.gz"
[13,] "fts" "D:/workspace/R/miniCRAN/pgk/fts_0.9.9.tar.gz"
[14,] "tis" "D:/workspace/R/miniCRAN/pgk/tis_1.34.tar.gz"
[15,] "RUnit" "D:/workspace/R/miniCRAN/pgk/RUnit_0.4.31.tar.gz"
本地目录查看下载的文件。
> dir("D:/workspace/R/miniCRAN/pgk")
[1] "BH_1.66.0-1.tar.gz" "chron_2.3-52.tar.gz" "curl_3.2.tar.gz"
[4] "fts_0.9.9.tar.gz" "lattice_0.20-35.tar.gz" "quadprog_1.5-5.tar.gz"
[7] "quantmod_0.4-13.tar.gz" "RUnit_0.4.31.tar.gz" "timeDate_3043.102.tar.gz"
[10] "timeSeries_3042.102.tar.gz" "tis_1.34.tar.gz" "tseries_0.10-44.tar.gz"
[13] "TTR_0.23-3.tar.gz" "xts_0.10-2.tar.gz" "zoo_1.8-1.tar.gz"
5.5 安装软件包
用R语言的函数,进行本地安装。
> install.packages(dp[,2], repos = NULL, type="source")
另外,我们也可以用命令行,进行安装。
> R CMD INSTALL D:\workspace\R\miniCRAN\pgk\zoo_1.8-1.tar.gz
通过使用miniCRAN包,就让我们找到依赖包的这事情,变得简单多了。再通过批量的离线下载,和批量的安装,就可以让我们的操作更有效率。不然,一个一包的安装,真是要把人给逼疯了!
虽然本文讲了通用的方法,但对于有一些包安装过程中,还需要从网上再下载其他的包,还需要具体问题具体分析,手动安装依赖包的过程,确实是无比复杂的。
转载请注明出处:
http://blog.fens.me/r-install-offline