R的极客理想系列文章,涵盖了R的思想,使用,工具,创新等的一系列要点,以我个人的学习和体验去诠释R的强大。
R语言作为统计学一门语言,一直在小众领域闪耀着光芒。直到大数据的爆发,R语言变成了一门炙手可热的数据分析的利器。随着越来越多的工程背景的人的加入,R语言的社区在迅速扩大成长。现在已不仅仅是统计领域,教育,银行,电商,互联网….都在使用R语言。
要成为有理想的极客,我们不能停留在语法上,要掌握牢固的数学,概率,统计知识,同时还要有创新精神,把R语言发挥到各个领域。让我们一起动起来吧,开始R的极客理想。
关于作者:
- 张丹(Conan), 程序员Java,R,PHP,Javascript
- weibo:@Conan_Z
- blog: http://blog.fens.me
- email: bsspirit@gmail.com
转载请注明出处:
http://blog.fens.me/r-data-table/
前言
在R语言中,我们最常用的数据类型是data.frame,绝大多数的数据处理的操作都是围绕着data.frame结构来做的。用data.frame可以很方便的进行数据存储和数据查询,配合apply族函数对数据循环计算,也可也用plyr, reshape2, melt等包对数据实现切分、分组、聚合等的操作。在数据量不太大的时候,使用起来很方便。但是,用data.frame结构处理数据时并不是很高效,特别是在稍大一点数据规模的时候,就会明显变慢。
data.table其实提供了一套和data.frame类似的功能,特别增加了索引的设置,让数据操作非常高效,可能会提升1-2数量级。本章就将data.table包的使用方法。
目录
- data.table包介绍
- data.table包的使用
- data.table包性能对比
1. data.table包介绍
data.table包是一个data.frame的扩展工具集,可以通过自定义keys来设置索引,实现高效的数据索引查询、快速分组、快速连接、快速赋值等数据操作。data.table主要通过二元检索法大大提高数据操作的效率,它也兼容适用于data.frame的向量检索法。同时,data.table对于大数据的快速聚合也有很好的效果,官方介绍说对于 100GB规模内存数据处理,运行效率还是很好的。那么,就让我们试验一下吧。
data.table项目地址:https://cran.r-project.org/web/packages/data.table/
本文所使用的系统环境
- Win10 64bit
- R: 3.2.3 x86_64-w64-mingw32/x64 b4bit
data.table包是在CRAN发布的标准库,安装起来非常简单,2条命令就可以了。
~ R
> install.packages("data.table")
> library(data.table)
2. data.table包的使用
接下来,开始用data.table包,并熟悉一下data.table包的基本操作。
2.1 用data.table创建数据集
通常情况,我们用data.frame创建一个数据集时,可以使用下面的语法。
# 创建一个data.frame数据框
> df<-data.frame(a=c('A','B','C','A','A','B'),b=rnorm(6))
> df
a b
1 A 1.3847248
2 B 0.6387315
3 C -1.8126626
4 A -0.0265709
5 A -0.3292935
6 B -1.0891958
对于data.table来说,创建一个数据集是和data.frame同样语法。
# 创建一个data.table对象
> dt = data.table(a=c('A','B','C','A','A','B'),b=rnorm(6))
> dt
a b
1: A 0.09174236
2: B -0.84029180
3: C -0.08157873
4: A -0.39992084
5: A -1.66034154
6: B -0.33526447
检查df, dt两个对象的类型,可以看到data.table是对data.frame的扩展类型。
# data.frame类型
> class(df)
[1] "data.frame"
# data.table类型
> class(dt)
[1] "data.table" "data.frame"
如果data.table仅仅是对data.frame的做了S3的扩展类型,那么data.table是不可能做到对data.frame从效率有极大的改进的。为了验证,我们需要检查一下data.table代码的结构定义。
# 打印data.table函数定义
> data.table
function (..., keep.rownames = FALSE, check.names = FALSE, key = NULL)
{
x <- list(...)
if (!.R.listCopiesNamed)
.Call(CcopyNamedInList, x)
if (identical(x, list(NULL)) || identical(x, list(list())) ||
identical(x, list(data.frame(NULL))) || identical(x,
list(data.table(NULL))))
return(null.data.table())
tt <- as.list(substitute(list(...)))[-1L]
vnames = names(tt)
if (is.null(vnames))
vnames = rep.int("", length(x))
vnames[is.na(vnames)] = ""
novname = vnames == ""
if (any(!novname)) {
if (any(vnames[!novname] == ".SD"))
stop("A column may not be called .SD. That has special meaning.")
}
for (i in which(novname)) {
if (is.null(ncol(x[[i]]))) {
if ((tmp <- deparse(tt[[i]])[1]) == make.names(tmp))
vnames[i] <- tmp
}
}
tt = vnames == ""
if (any(tt))
vnames[tt] = paste("V", which(tt), sep = "")
n <- length(x)
if (n < 1L)
return(null.data.table())
if (length(vnames) != n)
stop("logical error in vnames")
vnames <- as.list.default(vnames)
nrows = integer(n)
numcols = integer(n)
for (i in seq_len(n)) {
xi = x[[i]]
if (is.null(xi))
stop("column or argument ", i, " is NULL")
if ("POSIXlt" %chin% class(xi)) {
warning("POSIXlt column type detected and converted to POSIXct. We do not recommend use of POSIXlt at all because it uses 40 bytes to store one date.")
x[[i]] = as.POSIXct(xi)
}
else if (is.matrix(xi) || is.data.frame(xi)) {
xi = as.data.table(xi, keep.rownames = keep.rownames)
x[[i]] = xi
numcols[i] = length(xi)
}
else if (is.table(xi)) {
x[[i]] = xi = as.data.table.table(xi, keep.rownames = keep.rownames)
numcols[i] = length(xi)
}
nrows[i] <- NROW(xi)
if (numcols[i] > 0L) {
namesi <- names(xi)
if (length(namesi) == 0L)
namesi = rep.int("", ncol(xi))
namesi[is.na(namesi)] = ""
tt = namesi == ""
if (any(tt))
namesi[tt] = paste("V", which(tt), sep = "")
if (novname[i])
vnames[[i]] = namesi
else vnames[[i]] = paste(vnames[[i]], namesi, sep = ".")
}
}
nr <- max(nrows)
ckey = NULL
recycledkey = FALSE
for (i in seq_len(n)) {
xi = x[[i]]
if (is.data.table(xi) && haskey(xi)) {
if (nrows[i] < nr)
recycledkey = TRUE
else ckey = c(ckey, key(xi))
}
}
for (i in which(nrows < nr)) {
xi <- x[[i]]
if (identical(xi, list())) {
x[[i]] = vector("list", nr)
next
}
if (nrows[i] == 0L)
stop("Item ", i, " has no length. Provide at least one item (such as NA, NA_integer_ etc) to be repeated to match the ",
nr, " rows in the longest column. Or, all columns can be 0 length, for insert()ing rows into.")
if (nr%%nrows[i] != 0L)
warning("Item ", i, " is of size ", nrows[i], " but maximum size is ",
nr, " (recycled leaving remainder of ", nr%%nrows[i],
" items)")
if (is.data.frame(xi)) {
..i = rep(seq_len(nrow(xi)), length.out = nr)
x[[i]] = xi[..i, , drop = FALSE]
next
}
if (is.atomic(xi) || is.list(xi)) {
x[[i]] = rep(xi, length.out = nr)
next
}
stop("problem recycling column ", i, ", try a simpler type")
stop("argument ", i, " (nrow ", nrows[i], ") cannot be recycled without remainder to match longest nrow (",
nr, ")")
}
if (any(numcols > 0L)) {
value = vector("list", sum(pmax(numcols, 1L)))
k = 1L
for (i in seq_len(n)) {
if (is.list(x[[i]]) && !is.ff(x[[i]])) {
for (j in seq_len(length(x[[i]]))) {
value[[k]] = x[[i]][[j]]
k = k + 1L
}
}
else {
value[[k]] = x[[i]]
k = k + 1L
}
}
}
else {
value = x
}
vnames <- unlist(vnames)
if (check.names)
vnames <- make.names(vnames, unique = TRUE)
setattr(value, "names", vnames)
setattr(value, "row.names", .set_row_names(nr))
setattr(value, "class", c("data.table", "data.frame"))
if (!is.null(key)) {
if (!is.character(key))
stop("key argument of data.table() must be character")
if (length(key) == 1L) {
key = strsplit(key, split = ",")[[1L]]
}
setkeyv(value, key)
}
else {
if (length(ckey) && !recycledkey && !any(duplicated(ckey)) &&
all(ckey %in% names(value)) && !any(duplicated(names(value)[names(value) %in%
ckey])))
setattr(value, "sorted", ckey)
}
alloc.col(value)
}
<bytecode: 0x0000000017bfb990>
<environment: namespace:data.table>
从上面的整个大段代码来看,data.table的代码定义中并没有使用data.frame结构的依赖的代码,data.table都在自己函数定义中做的数据处理,所以我们可以确认data.table和data.frame的底层结果是不一样的。
那么为什么从刚刚用class函数检查data.table对象时,会看到data.table和data.frame的扩展关系呢?这里就要了解R语言中对于S3面向对象系统的结构设计了,关于S3的面向对象设计,请参考文章R语言基于S3的面向对象编程。
从上面代码中,倒数第17行找到 setattr(value, "class", c("data.table", "data.frame")) 这行,发现这个扩展的定义是作者主动设计的,那么其实就可以理解为,data.table包的作者希望data.table使用起来更像data.frame,所以通过一些包装让使用者无切换成本的。
2.2 data.table和data.frame相互转换
如果想把data.frame对象和data.table对象进行转换,转换的代码是非常容易的,直接转换就可以了。
从一个data.frame对象转型到data.table对象。
# 创建一个data.frame对象
> df<-data.frame(a=c('A','B','C','A','A','B'),b=rnorm(6))
# 检查类型
> class(df)
[1] "data.frame"
# 转型为data.table对象
> df2<-data.table(df)
# 检查类型
> class(df2)
[1] "data.table" "data.frame"
从一个data.table对象转型到data.frame对象。
# 创建一个data.table对象
> dt <- data.table(a=c('A','B','C','A','A','B'),b=rnorm(6))
# 检查类型
> class(dt)
[1] "data.table" "data.frame"
# 转型为data.frame对象
> dt2<-data.frame(dt)
# 检查类型
> class(dt2)
[1] "data.frame"
2.3 用data.table进行查询
由于data.table对用户使用上是希望和data.frame的操作尽量相似,所以适用于data.frame的查询方法基本都适用于data.table,同时data.table自己具有的一些特性,提供了自定义keys来进行高效的查询。
下面先看一下,data.table基本的数据查义方法。
# 创建一个data.table对象
> dt = data.table(a=c('A','B','C','A','A','B'),b=rnorm(6))
> dt
a b
1: A 0.7792728
2: B 1.4870693
3: C 0.9890549
4: A -0.2769280
5: A -1.3009561
6: B 1.1076424
按行或按列查询
# 取第二行的数据
> dt[2,]
a b
1: B 1.487069
# 不加,也可以
> dt[2]
a b
1: B 1.487069
# 取a列的值
> dt$a
[1] "A" "B" "C" "A" "A" "B"
# 取a列中值为B的行
> dt[a=="B",]
a b
1: B 1.487069
2: B 1.107642
# 取a列中值为B的行的判断
> dt[,a=='B']
[1] FALSE TRUE FALSE FALSE FALSE TRUE
# 取a列中值为B的行的索引
> which(dt[,a=='B'])
[1] 2 6
上面的操作,不管是用索引值,== 和 $ 都是data.frame操作一样的。下面我们取data.table特殊设计的keys来查询。
# 设置a列为索引列
> setkey(dt,a)
# 打印dt对象,发现数据已经按照a列字母对应ASCII码值进行了排序。
> dt
a b
1: A 0.7792728
2: A -0.2769280
3: A -1.3009561
4: B 1.4870693
5: B 1.1076424
6: C 0.9890549
按照自定义的索引进行查询。
# 取a列中值为B的行
> dt["B",]
a b
1: B 1.487069
2: B 1.107642
# 取a列中值为B的行,并保留第一行
> dt["B",mult="first"]
a b
1: B 1.487069
# 取a列中值为B的行,并保留最后一行
> dt["B",mult="last"]
a b
1: B 1.107642
# 取a列中值为b的行,没有数据则为NA
> dt["b"]
a b
1: b NA
从上面的代码测试中我们可以看出,在定义了keys后,我们要查询的时候就不用再指定列了,默认会把方括号中的第一位置留给keys,作为索引匹配的查询条件。从代码的角度,又节省了一个变量定义的代码。同时,可以用mult参数,对数据集增加过滤条件,让代码本身也变得更高效。如果查询的值,不是索引列包括的值,则返回NA。
2.4 对data.table对象进行增、删、改操作
给data.table对象增加一列,可以使用这样的格式 data.table[, colname := var1]。
# 创建data.table对象
> dt = data.table(a=c('A','B','C','A','A','B'),b=rnorm(6))
> dt
a b
1: A 1.51765578
2: B 0.01182553
3: C 0.71768667
4: A 0.64578235
5: A -0.04210508
6: B 0.29767383
# 增加1列,列名为c
> dt[,c:=b+2]
> dt
a b c
1: A 1.51765578 3.517656
2: B 0.01182553 2.011826
3: C 0.71768667 2.717687
4: A 0.64578235 2.645782
5: A -0.04210508 1.957895
6: B 0.29767383 2.297674
# 增加2列,列名为c1,c2
> dt[,`:=`(c1 = 1:6, c2 = 2:7)]
> dt
a b c c1 c2
1: A 0.7545555 2.754555 1 2
2: B 0.5556030 2.555603 2 3
3: C -0.1080962 1.891904 3 4
4: A 0.3983576 2.398358 4 5
5: A -0.9141015 1.085899 5 6
6: B -0.8577402 1.142260 6 7
# 增加2列,第2种写法
> dt[,c('d1','d2'):=list(1:6,2:7)]
> dt
a b c c1 c2 d1 d2
1: A 0.7545555 2.754555 1 2 1 2
2: B 0.5556030 2.555603 2 3 2 3
3: C -0.1080962 1.891904 3 4 3 4
4: A 0.3983576 2.398358 4 5 4 5
5: A -0.9141015 1.085899 5 6 5 6
6: B -0.8577402 1.142260 6 7 6 7
给data.table对象删除一列时,就是给这列赋值为空,使用这样的格式 data.table[, colname := NULL]。我们继续使用刚才创建的dt对象。
# 删除c1列
> dt[,c1:=NULL]
> dt
a b c c2 d1 d2
1: A 0.7545555 2.754555 2 1 2
2: B 0.5556030 2.555603 3 2 3
3: C -0.1080962 1.891904 4 3 4
4: A 0.3983576 2.398358 5 4 5
5: A -0.9141015 1.085899 6 5 6
6: B -0.8577402 1.142260 7 6 7
# 同时删除d1,d2列
> dt[,c('d1','d2'):=NULL]
> dt
a b c c2
1: A 0.7545555 2.754555 2
2: B 0.5556030 2.555603 3
3: C -0.1080962 1.891904 4
4: A 0.3983576 2.398358 5
5: A -0.9141015 1.085899 6
6: B -0.8577402 1.142260 7
修改data.table对象的值,就是通过索引定位后进行值的替换,通过这样的格式 data.table[condition, colname := 0]。我们继续使用刚才创建的dt对象。
# 给b赋值为30
> dt[,b:=30]
> dt
a b c c2
1: A 30 2.754555 2
2: B 30 2.555603 3
3: C 30 1.891904 4
4: A 30 2.398358 5
5: A 30 1.085899 6
6: B 30 1.142260 7
# 对a列值为B的行,c2列值值大于3的行,的b列赋值为100
> dt[a=='B' & c2>3, b:=100]
> dt
a b c c2
1: A 30 2.754555 2
2: B 30 2.555603 3
3: C 30 1.891904 4
4: A 30 2.398358 5
5: A 30 1.085899 6
6: B 100 1.142260 7
# 还有另一种写法
> dt[,b:=ifelse(a=='B' & c2>3,50,b)]
> dt
a b c c2
1: A 30 2.754555 2
2: B 30 2.555603 3
3: C 30 1.891904 4
4: A 30 2.398358 5
5: A 30 1.085899 6
6: B 50 1.142260 7
2.5 data.table的分组计算
基于data.frame对象做分组计算时,要么使用apply函数自己处理,要么用plyr包的分组计算功能。对于data.table包本身就支持了分组计算,很像SQL的group by这样的功能,这是data.table包主打的优势。
比如,按a列分组,并对b列按分组求和。
# 创建数据
> dt = data.table(a=c('A','B','C','A','A','B'),b=rnorm(6))
> dt
a b
1: A 1.4781041
2: B 1.4135736
3: C -0.6593834
4: A -0.1231766
5: A -1.7351749
6: B -0.2528973
# 对整个b列数据求和
> dt[,sum(b)]
[1] 0.1210455
# 按a列分组,并对b列按分组求和
> dt[,sum(b),by=a]
a V1
1: A -0.3802474
2: B 1.1606763
3: C -0.6593834
2.6 多个data.table的连接操作
在操作数据的时候,经常会出现2个或多个数据集通过一个索引键进行关联,而我们的算法要把多种数据合并到一起再进行处理,那么这个时候就会用的数据的连接操作,类似关系型数据库的左连接(LEFT JOIN)。
举个例子,学生考试的场景。按照ER设计方法,我们通常会按照实体进行数据划分。这里存在2个实体,一个是学生,一个是成绩。学生实体会包括,学生姓名等的基本资料,而成绩实体会包括,考试的科目,考试的成绩。
假设有6个学生,分别参加A和B两门考试,每门考试得分是不一样的。
# 6个学生
> student <- data.table(id=1:6,name=c('Dan','Mike','Ann','Yang','Li','Kate'));student
id name
1: 1 Dan
2: 2 Mike
3: 3 Ann
4: 4 Yang
5: 5 Li
6: 6 Kate
# 分别参加A和B两门考试
> score <- data.table(id=1:12,stuId=rep(1:6,2),score=runif(12,60,99),class=c(rep('A',6),rep('B',6)));score
id stuId score class
1: 1 1 89.18497 A
2: 2 2 61.76987 A
3: 3 3 74.67598 A
4: 4 4 64.08165 A
5: 5 5 85.00035 A
6: 6 6 95.25072 A
7: 7 1 81.42813 B
8: 8 2 82.16083 B
9: 9 3 69.53405 B
10: 10 4 89.01985 B
11: 11 5 96.77196 B
12: 12 6 97.02833 B
通过学生ID,把学生和考试成绩2个数据集进行连接。
# 设置score数据集,key为stuId
> setkey(score,"stuId")
# 设置student数据集,key为id
> setkey(student,"id")
# 合并两个数据集的数据
> student[score,nomatch=NA,mult="all"]
id name i.id score class
1: 1 Dan 1 89.18497 A
2: 1 Dan 7 81.42813 B
3: 2 Mike 2 61.76987 A
4: 2 Mike 8 82.16083 B
5: 3 Ann 3 74.67598 A
6: 3 Ann 9 69.53405 B
7: 4 Yang 4 64.08165 A
8: 4 Yang 10 89.01985 B
9: 5 Li 5 85.00035 A
10: 5 Li 11 96.77196 B
11: 6 Kate 6 95.25072 A
12: 6 Kate 12 97.02833 B
最后我们会看到,两个数据集的结果合并在了一个结果数据集中。这样就完成了,数据连接的操作。从代码的角度来看,1行代码要比用data.frame去拼接方便的多。
3. data.table包性能对比
现在很多时候我们需要处理的数据量是很大的,动辄上百万行甚至上千万行。如果我们要使用R对其进行分析或处理,在不增加硬件的条件下,就需要用一些高性能的数据包进行数据的操作。这里就会发现data.table是非常不错的一个选择。
3.1 data.table和data.frame索引查询性能对比
我们先生成一个稍大数据集,包括2列x和y分别用英文字母进行赋值,100,000,004行,占内存大小1.6G。分别比较data.frame操作和data.table操作的索引查询性能耗时。
使用data.frame创建数据集。
# 清空环境变量
> rm(list=ls())
# 设置大小
> size = ceiling(1e8/26^2)
[1] 147929
# 计算data.frame对象生成的时间
> t0=system.time(
+ df <- data.frame(x=rep(LETTERS,each=26*size),y=rep(letters,each=size))
+ )
# 打印时间
> t0
用户 系统 流逝
3.63 0.18 3.80
# df对象的行数
> nrow(df)
[1] 100000004
# 占用内存
> object.size(df)
1600003336 bytes
# 进行条件查询
> t1=system.time(
+ val1 <- dt[dt$x=="R" & dt$y=="h",]
+ )
# 查询时间
> t1
用户 系统 流逝
8.53 0.84 9.42
再使用data.table创建数据集。
# 清空环境变量
> rm(list=ls())
# 设置大小
> size = ceiling(1e8/26^2)
[1] 147929
# 计算data.table对象生成的时间
> t3=system.time(
+ dt <- data.table(x=rep(LETTERS,each=26*size),y=rep(letters,each=size))
+ )
# 生成对象的时间
> t3
用户 系统 流逝
3.22 0.39 3.63
# 对象行数
> nrow(dt)
[1] 100000004
# 占用内存
> object.size(dt)
2000004040 bytes
# 进行条件查询
> t3=system.time(
+ val2 <- dt[x=="R" & y=="h",]
+ )
# 查询时间
> t3
用户 系统 流逝
6.52 0.26 6.80
从上面的测试来看,创建对象时,data.table比data.frame显著的高效,而查询效果则并不明显。我们对data.table数据集设置索引,试试有索引查询的效果。
# 设置key索引列为x,y
> setkey(dt,x,y)
# 条件查询
> t4=system.time(
+ val3 <- dt[list("R","h")]
+ )
# 查看时间
> t4
用户 系统 流逝
0.00 0.00 0.06
设置索引列后,按索引进行查询,无CPU耗时。震惊了!!
3.2 data.table和data.frame的赋值性能对比
对于赋值操作来说,通常会分为2个动作,先查询再值替换,对于data.frame和data.table都是会按照这个过程来实现的。从上一小节中,可以看到通过索引查询时data.table比data.frame明显的速度要快,对于赋值的操作测试,我们就要最好避免复杂的查询。
对x列值为R的行,对应的y的值进行赋值。首先测试data.frame的计算时间。
> size = 1000000
> df <- data.frame(x=rep(LETTERS,each=size),y=rnorm(26*size))
> system.time(
+ df$y[which(df$x=='R')]<-10
+ )
用户 系统 流逝
0.75 0.01 0.77
计算data.table的赋值时间。
> dt <- data.table(x=rep(LETTERS,each=size),y=rnorm(26*size))
> system.time(
+ dt[x=='R', y:=10]
+ )
用户 系统 流逝
0.11 0.00 0.11
> setkey(dt,x)
> system.time(
+ dt['R', y:=10]
+ )
用户 系统 流逝
0.01 0.00 0.02
通过对比data.table和data.frame的赋值测试,有索引的data.table性能优势是非常明显的。我们增大数据量,再做一次赋值测试。
> size = 1000000*5
> df <- data.frame(x=rep(LETTERS,each=size),y=rnorm(26*size))
> system.time(
+ df$y[which(df$x=='R')]<-10
+ )
用户 系统 流逝
3.22 0.25 3.47
> rm(list=ls())
> size = 1000000*5
> dt <- data.table(x=rep(LETTERS,each=size),y=rnorm(26*size))
> setkey(dt,x)
> system.time(
+ dt['R', y:=10]
+ )
用户 系统 流逝
0.08 0.01 0.08
对于增加数据量后data.table,要比data.frame的赋值快更多倍。
3.3 data.table和tapply分组计算性能对比
再对比一下data.table处理数据和tapply的分组计算的性能。测试同样地只做一个简单的计算设定,比如,对一个数据集按x列分组对y列求和。
# 设置数据集大小
> size = 100000
> dt <- data.table(x=rep(LETTERS,each=size),y=rnorm(26*size))
# 设置key为x列
> setkey(dt,x)
# 计算按x列分组,对y列的求和时间
> system.time(
+ r1<-dt[,sum(y),by=x]
+ )
用户 系统 流逝
0.03 0.00 0.03
# 用tapply实现,计算求和时间
> system.time(
+ r2<-tapply(dt$y,dt$x,sum)
+ )
用户 系统 流逝
0.25 0.05 0.30
# 查看数据集大小, 40mb
> object.size(dt)
41602688 bytes
对于40mb左右的数据来说,tapply比data.table要快,那么我增加数据集的大小,给size*10再测试一下。
> size = 100000*10
> dt <- data.table(x=rep(LETTERS,each=size),y=rnorm(26*size))
> setkey(dt,x)
> val3<-dt[list("R")]
> system.time(
+ r1<-dt[,sum(y),by=x]
+ )
用户 系统 流逝
0.25 0.03 0.28
> system.time(
+ r2<-tapply(dt$y,dt$x,sum)
+ )
用户 系统 流逝
2.56 0.36 2.92
# 400mb数据
> object.size(dt)
416002688 bytes
对于400mb的数据来说,data.table的计算性能已经明显优于tapply了,再把数据时增加让size*5。
> size = 100000*10*5
> dt <- data.table(x=rep(LETTERS,each=size),y=rnorm(26*size))
> setkey(dt,x)
> system.time(
+ r1<-dt[,sum(y),by=x]
+ )
用户 系统 流逝
1.50 0.11 1.61
> system.time(
+ r2<-tapply(dt$y,dt$x,sum)
+ )
用户 系统 流逝
13.30 3.58 16.90
# 2G数据
> object.size(dt)
2080002688 bytes
对于2G左右的数据来说,tapply总耗时到了16秒,而data.table为1.6秒,从2个的测试来说,大于400mb数据时CPU耗时是线性的。
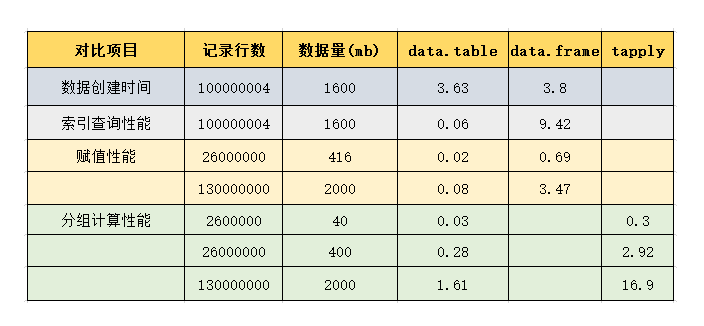
把上几组测试数据放到一起,下图所示。
通过上面的对比,我们发现data.table包比tapply快10倍,比data.frame赋值操作快30倍,比data.frame的索引查询快100倍,绝对是值得花精力去学习的一个包。
赶紧用data.table包去优化你的程序吧!
转载请注明出处:
http://blog.fens.me/r-data-table/