R的极客理想系列文章,涵盖了R的思想,使用,工具,创新等的一系列要点,以我个人的学习和体验去诠释R的强大。
R语言作为统计学一门语言,一直在小众领域闪耀着光芒。直到大数据的爆发,R语言变成了一门炙手可热的数据分析的利器。随着越来越多的工程背景的人的加入,R语言的社区在迅速扩大成长。现在已不仅仅是统计领域,教育,银行,电商,互联网….都在使用R语言。
要成为有理想的极客,我们不能停留在语法上,要掌握牢固的数学,概率,统计知识,同时还要有创新精神,把R语言发挥到各个领域。让我们一起动起来吧,开始R的极客理想。
关于作者:
- 张丹, 程序员R,Nodejs,Java
- weibo:@Conan_Z
- blog: http://blog.fens.me
- email: bsspirit@gmail.com
转载请注明出处:
http://blog.fens.me/r-tibble/
前言
最近正在整理用R语言进行数据处理的操作方法,发现了 RStudio 公司开发的数据科学工具包tidyverse,一下子就把我吸引了。通过2天时间,我把tidyverse项目整体的学了一遍,给我的启发是非常大的。tidyverse 重新定义了数据科学的工作路径,而且路径上每个核心节点,都定义了对应的R包。这真是一项造福数据分析行业的工程,非常值得称赞!!
tidyverse个项目,包括了一系列的子项目,其中tibble被定义为取代传统data.frame的数据类型,完全有颠覆R的数据操作的可能。跟上R语言领袖的脚步,领先进入数据科学新的时代。
目录
- tibble介绍
- tibble安装
- tibble包的基本使用
- tibble的源代码分析
1. tibble介绍
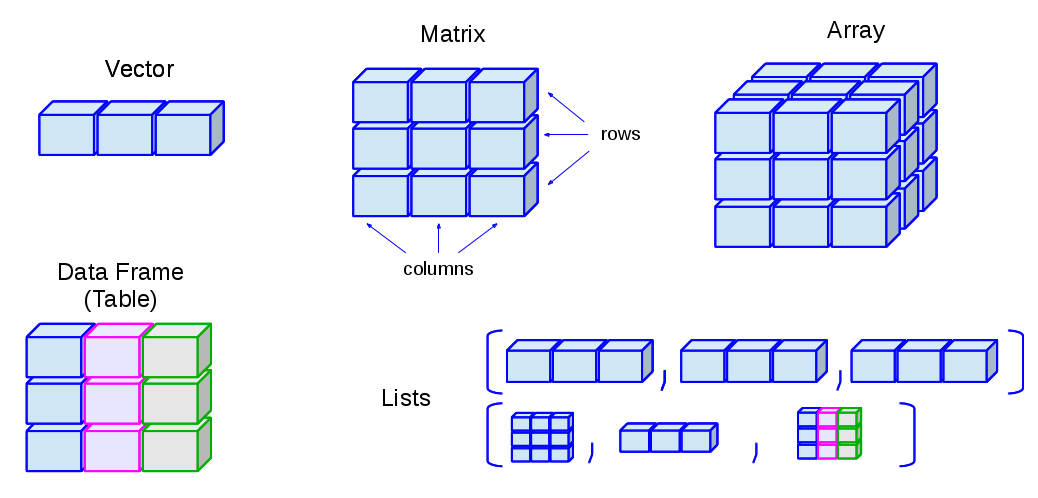
tibble是R语言中一个用来替换data.frame类型的扩展的数据框,tibble继承了data.frame,是弱类型的,同时与data.frame有相同的语法,使用起来更方便。tibble包,也是由Hadley开发的R包。
tibble对data.frame做了重新的设定:
- tibble,不关心输入类型,可存储任意类型,包括list类型
- tibble,没有行名设置 row.names
- tibble,支持任意的列名
- tibble,会自动添加列名
- tibble,类型只能回收长度为1的输入
- tibble,会懒加载参数,并按顺序运行
- tibble,是tbl_df类型
tibble的项目主页:https://github.com/tidyverse/tibble
2. tibble安装
本文所使用的系统环境
- Win10 64bit
- R: 3.2.3 x86_64-w64-mingw32/x64 b4bit
tibble是在CRAN发布的标准库,安装起来非常简单,2条命令就可以了。
~ R
> install.packages('tibble')
> library(tibble)
RStudio官方把tibble项目,集成到了tidyverse项目中了,官方建议直接安装tidyverse项目,这样整个用来做数据科学的库都会被下载下来。
~ R
> install.packages('tidyverse')
> library(tidyverse)
#> Loading tidyverse: ggplot2
#> Loading tidyverse: tibble
#> Loading tidyverse: tidyr
#> Loading tidyverse: readr
#> Loading tidyverse: purrr
#> Loading tidyverse: dplyr
#> Conflicts with tidy packages ----------------------------------------------
#> filter(): dplyr, stats
#> lag(): dplyr, stats
tidyverse项目,是一个包括了数据科学的一个集合工具项目,用于数据提取,数据清理,数据类型定义,数据处理,数据建模,函数化编程,数据可视化,包括了下面的包。
- ggplot2, 数据可视化
- dplyr, 数据处理
- tidyr, 数据清理
- readr, 数据提取
- purrr, 函数化编程
- tibble, 数据类型定义
tidyverse项目的地址:https://github.com/tidyverse/tidyverse。高效的使用R语言做数据科学,请参考开源图书 R for Data Science.
3. tibble包的基本使用
对于tibble包的使用,主要需要掌握创建、数据转型、数据查看、数据操作、与data.frame的区别点。复杂的数据处理功能,是dplyr项目来完成,下一篇讲dplyr的文章再给大家介绍。
3.1 创建tibble
创建一个tibble类型的data.frame是非常简单的,语法与传统的data.frame是类似的。
# 创建一个tibble类型的data.frame
> t1<-tibble(1:10,b=LETTERS[1:10]);t1
# A tibble: 10 x 2
`1:10` b
<int> <chr>
1 1 A
2 2 B
3 3 C
4 4 D
5 5 E
6 6 F
7 7 G
8 8 H
9 9 I
10 10 J
# 创建一个data.frame
> d1<-data.frame(1:10,b=LETTERS[1:10]);d1
X1.10 b
1 1 A
2 2 B
3 3 C
4 4 D
5 5 E
6 6 F
7 7 G
8 8 H
9 9 I
10 10 J
从上面的输出可以看到tibble类型,会在输出时多一行,用来指定每一列的类型。
tibble用缩写定义了7种类型:
- int,代表integer
- dbl,代表double
- chr,代表character向量或字符串。
- dttm,代表日期+时间(a date + a time)
- lgl,代表逻辑判断TRUE或者FALSE
- fctr,代表因子类型factor
- date,代表日期dates.
查看类型,发现tbl_df继承了tbl继承是data.frame,所以tibble是data.frame的子类型。
# t1为tbl_df类型
> class(t1)
[1] "tbl_df" "tbl" "data.frame"
# 是data.frame类型
> class(d1)
[1] "data.frame"
让我们多角度来观察t1变量。
# 判断是不是tibble类型
> is.tibble(t1)
[1] TRUE
# 查看t1的属性
> attributes(t1)
$names
[1] "1:10" "b"
$class
[1] "tbl_df" "tbl" "data.frame"
$row.names
[1] 1 2 3 4 5 6 7 8 9 10
# 查看t1的静态结构
> str(t1)
Classes ‘tbl_df’, ‘tbl’ and 'data.frame': 10 obs. of 2 variables:
$ 1:10: int 1 2 3 4 5 6 7 8 9 10
$ b : chr "A" "B" "C" "D" ...
通过文本排列来创建一个tibble
> tribble(
+ ~colA, ~colB,
+ "a", 1,
+ "b", 2,
+ "c", 3
+ )
# A tibble: 3 x 2
colA colB
<chr> <dbl>
1 a 1
2 b 2
3 c 3
通过vector创建tibble
> tibble(letters)
# A tibble: 26 x 1
letters
<chr>
1 a
2 b
3 c
4 d
5 e
6 f
7 g
8 h
9 i
10 j
# ... with 16 more rows
通过data.frame创建tibble,这时就会报错了。
> tibble(data.frame(1:5))
Error: Column `data.frame(1:5)` must be a 1d atomic vector or a list
通过list创建tibble
> tibble(x = list(diag(1), diag(2)))
# A tibble: 2 x 1
x
<list>
1 <dbl [1 x 1]>
2 <dbl [2 x 2]>
我们看到tibble其实是存储list类型,这是data.frame做不到的。
通过一个tibble,创建另一个tibble,这时也会报错了。
> tibble(x = tibble(1, 2, 3))
Error: Column `x` must be a 1d atomic vector or a list
3.2 数据类型转换
tibble是一个新的类型,R语言中大部分的数据都是基于原有的数据类型,所以原有数据类型与tiblle类型的转换就显的非常重要了。
把一个data.frame的类型的转换为tibble类型
# 定义一个data.frame类型变量
> d1<-data.frame(1:5,b=LETTERS[1:5]);d1
X1.5 b
1 1 A
2 2 B
3 3 C
4 4 D
5 5 E
# 把data.frame转型为tibble
> d2<-as.tibble(d1);d2
# A tibble: 5 x 2
X1.5 b
<int> <fctr>
1 1 A
2 2 B
3 3 C
4 4 D
5 5 E
# 再转回data.frame
> as.data.frame(d2)
X1.5 b
1 1 A
2 2 B
3 3 C
4 4 D
5 5 E
我们可以看到tibble与data.frame的转型是非常平滑的,一个转型函数就够,不需要中间做任何的特殊处理。
把一个vector转型为tibble类型,但是不能再转回vector了。
# vector转型到tibble
> x<-as.tibble(1:5);x
# A tibble: 5 x 1
value
<int>
1 1
2 2
3 3
4 4
5 5
# tibble转型到vector, 不成功
> as.vector(x)
# A tibble: 5 x 1
value
<int>
1 1
2 2
3 3
4 4
5 5
把list转型为tibble。
# 把list转型为tibble
> df <- as.tibble(list(x = 1:500, y = runif(500), z = 500:1));df
# A tibble: 500 x 3
x y z
<int> <dbl> <int>
1 1 0.59141749 500
2 2 0.61926125 499
3 3 0.06879729 498
4 4 0.69579561 497
5 5 0.05087461 496
6 6 0.63172517 495
7 7 0.41808985 494
8 8 0.78110219 493
9 9 0.95279741 492
10 10 0.98930640 491
# ... with 490 more rows
# 把tibble再转为list
> str(as.list(df))
List of 3
$ x: int [1:500] 1 2 3 4 5 6 7 8 9 10 ...
$ y: num [1:500] 0.5914 0.6193 0.0688 0.6958 0.0509 ...
$ z: int [1:500] 500 499 498 497 496 495 494 493 492 491 ...
tibble与list的转型也是非常平滑的,一个转型函数就够。
把matrix转型为tibble。
# 生成一个matrix
> m <- matrix(rnorm(15), ncol = 5)
# matrix转为tibble
> df <- as.tibble(m);df
# A tibble: 3 x 5
V1 V2 V3 V4 V5
1 0.8436494 2.1420238 0.2690392 -0.4752708 -0.2334994
2 1.0363340 0.8653771 -0.3200777 -1.7400856 1.2253651
3 -0.2170344 -1.1346455 0.2204718 1.2189431 0.7020156
# tibble转为matrix
> as.matrix(df)
V1 V2 V3 V4 V5
[1,] 0.8436494 2.1420238 0.2690392 -0.4752708 -0.2334994
[2,] 1.0363340 0.8653771 -0.3200777 -1.7400856 1.2253651
[3,] -0.2170344 -1.1346455 0.2204718 1.2189431 0.7020156
从上面的转型测试可以看到,tibble类型是非常友好的,可以与data.frame, list, matrix 进行相互转型操作。tibble与vector是不能进行直接转型的,这与data.frame的行为是一致的,如果需要转型,我们可以分别取出每一列进行拼接,或转为matrix再操作。
3.3 tibble数据查询
通常我们是str()函数来观察数据的静态组成结果,在tibble包提供了一个glimpse(),可以方便我们来观察tibble和data.frame类型的数据。
比较glimpse()和str()对于data.frame的数据查看输出
> glimpse(mtcars)
Observations: 32
Variables: 11
$ mpg 21.0, 21.0, 22.8, 21.4, 18.7, 18.1, 14.3, 24.4, 22.8, 19.2, 17.8, 16.4, 17....
$ cyl 6, 6, 4, 6, 8, 6, 8, 4, 4, 6, 6, 8, 8, 8, 8, 8, 8, 4, 4, 4, 4, 8, 8, 8, 8, ...
$ disp 160.0, 160.0, 108.0, 258.0, 360.0, 225.0, 360.0, 146.7, 140.8, 167.6, 167.6...
$ hp 110, 110, 93, 110, 175, 105, 245, 62, 95, 123, 123, 180, 180, 180, 205, 215...
$ drat 3.90, 3.90, 3.85, 3.08, 3.15, 2.76, 3.21, 3.69, 3.92, 3.92, 3.92, 3.07, 3.0...
$ wt 2.620, 2.875, 2.320, 3.215, 3.440, 3.460, 3.570, 3.190, 3.150, 3.440, 3.440...
$ qsec 16.46, 17.02, 18.61, 19.44, 17.02, 20.22, 15.84, 20.00, 22.90, 18.30, 18.90...
$ vs 0, 0, 1, 1, 0, 1, 0, 1, 1, 1, 1, 0, 0, 0, 0, 0, 0, 1, 1, 1, 1, 0, 0, 0, 0, ...
$ am 1, 1, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 1, 0, 0, 0, 0, 0, ...
$ gear 4, 4, 4, 3, 3, 3, 3, 4, 4, 4, 4, 3, 3, 3, 3, 3, 3, 4, 4, 4, 3, 3, 3, 3, 3, ...
$ carb 4, 4, 1, 1, 2, 1, 4, 2, 2, 4, 4, 3, 3, 3, 4, 4, 4, 1, 2, 1, 1, 2, 2, 4, 2, ...
# 打印静态结构
> str(mtcars)
'data.frame': 32 obs. of 11 variables:
$ mpg : num 21 21 22.8 21.4 18.7 18.1 14.3 24.4 22.8 19.2 ...
$ cyl : num 6 6 4 6 8 6 8 4 4 6 ...
$ disp: num 160 160 108 258 360 ...
$ hp : num 110 110 93 110 175 105 245 62 95 123 ...
$ drat: num 3.9 3.9 3.85 3.08 3.15 2.76 3.21 3.69 3.92 3.92 ...
$ wt : num 2.62 2.88 2.32 3.21 3.44 ...
$ qsec: num 16.5 17 18.6 19.4 17 ...
$ vs : num 0 0 1 1 0 1 0 1 1 1 ...
$ am : num 1 1 1 0 0 0 0 0 0 0 ...
$ gear: num 4 4 4 3 3 3 3 4 4 4 ...
$ carb: num 4 4 1 1 2 1 4 2 2 4 ...
比较glimpse()和str()对于tibble的数据查看输出。
# 新建tibble
> df <- tibble(x = rnorm(500), y = rep(LETTERS[1:25],20))
# 查看df
> glimpse(df)
Observations: 500
Variables: 2
$ x -0.3295530, -2.0440424, 0.1444697, 0.8752439, 1.7705952, 0.5898253, 0.1991844,...
$ y "A", "B", "C", "D", "E", "F", "G", "H", "I", "J", "K", "L", "M", "N", "O", "P"...
# 查看df静态结构
> str(df)
Classes ‘tbl_df’, ‘tbl’ and 'data.frame': 500 obs. of 2 variables:
$ x: num -0.33 -2.044 0.144 0.875 1.771 ...
$ y: chr "A" "B" "C" "D" ...
按列出数据,一层[]返回的结果还是tibbe,二层[]与$返回的结果为列组成的向量。
> df <- tibble(x = 1:3, y = 3:1)
# 按列取,返回tibble
> df[1]
# A tibble: 3 x 1
x
<int>
1 1
2 2
3 3
# 按列取,返回向量
> df[[1]]
[1] 1 2 3
> df$x
[1] 1 2 3
按行取数据,这时一定要用,来做分隔符
# 取第一行
> df[1,]
# A tibble: 1 x 2
x y
<int> <int>
1 1 3
# 取前2行
> df[1:2,]
# A tibble: 2 x 2
x y
<int> <int>
1 1 3
2 2 2
# 取第二列的2,3行
> df[2:3,2]
# A tibble: 2 x 1
y
<int>
1 2
2 1
3.4 tibble数据操作
增加一列。
# 创建一个tibble
> df <- tibble(x = 1:3, y = 3:1);df
# A tibble: 3 x 2
x y
<int> <int>
1 1 3
2 2 2
3 3 1
# 增加一列
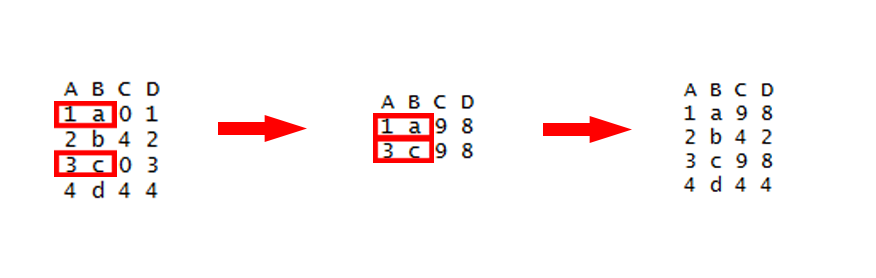
> add_column(df, z = -1:1, w = 0)
# A tibble: 3 x 4
x y z w
<int> <int> <int> <dbl>
1 1 3 -1 0
2 2 2 0 0
3 3 1 1 0
增加一行,还是基于上面生成的df变量。
# 在最后,增加一行
> add_row(df, x = 99, y = 9)
# A tibble: 4 x 2
x y
<dbl> <dbl>
1 1 3
2 2 2
3 3 1
4 99 9
# 插入第二行,增加一行
> add_row(df, x = 99, y = 9, .before = 2)
# A tibble: 4 x 2
x y
<dbl> <dbl>
1 1 3
2 99 9
3 2 2
4 3 1
3.5 tibble与data.frame的区别
列名,可以自由定义,并且会自动补全。
> tb <- tibble(
+ `:)` = "smile",
+ ` ` = "space",
+ `2000` = "number",
+ `列名` = "hi",
+ 1,1L
+ )
> tb
# A tibble: 1 x 6
`:)` ` ` `2000` 列名 `1` `1L`
<chr> <chr> <chr> <chr> <dbl> <int>
1 smile space number hi 1 1
数据,按顺序执行懒加载。
> a <- 1:5
> tibble(a, b = a * 2)
# A tibble: 5 x 2
a b
<int> <dbl>
1 1 2
2 2 4
3 3 6
4 4 8
5 5 10
打印输出控制,tibble的打印控制被重写了,所以执行print()函数时,模型会先进行类型匹配,然后调用print.tbl()。
# 创建tiblle
> tb<-tibble(a=1:5, b = a * 2, c=NA, d='a', e=letters[1:5])
# 打印前10行,不限宽度
> print(tb,n = 10, width = Inf)
# A tibble: 5 x 5
a b c d e
<int> <dbl> <lgl> <chr> <chr>
1 1 2 NA a a
2 2 4 NA a b
3 3 6 NA a c
4 4 8 NA a d
5 5 10 NA a e
# 打印前3行,宽度30
> print(tb,n = 3, width = 30)
# A tibble: 5 x 5
a b c d
<int> <dbl> <lgl> <chr>
1 1 2 NA a
2 2 4 NA a
3 3 6 NA a
# ... with 2 more rows, and 1
# more variables: e
# 用print函数,打印data.frame
> df<-data.frame(tb)
> print(df)
a b c d e
1 1 2 NA a a
2 2 4 NA a b
3 3 6 NA a c
4 4 8 NA a d
5 5 10 NA a e
3.7 特殊的函数
lst,创建一个list,具有tibble特性的list。 lst函数的工作原理,类似于执行[list()],这样的操作。
# 创建一个list,懒加载,顺序执行
> lst(n = 5, x = runif(n))
$n
[1] 5
$x
[1] 0.6417069 0.2674489 0.5610810 0.1771051 0.1504583
enframe,快速创建tibble。enframe提供了一个模板,只有2列name和value,快速地把2个向量匹配的tibble中,可以按行生成或按列生成。
# 按列生成
> enframe(1:3)
# A tibble: 3 x 2
name value
<int> <int>
1 1 1
2 2 2
3 3 3
# 按行生成
> enframe(c(a = 5, b = 7))
# A tibble: 2 x 2
name value
<chr> <dbl>
1 a 5
2 b 7
deframe,把tibble反向转成向量,这个函数就实现了,tibble到向量的转换。它默认把name列为索引,用value为值。
# 生成tibble
> df<-enframe(c(a = 5, b = 7));df
# A tibble: 2 x 2
name value
<chr> <dbl>
1 a 5
2 b 7
# 转为vector
> deframe(df)
a b
5 7
3.8 用于处理data.frame函数
tibble还提供了一些用于处理data.frame的函数。
# 创建data.frame
> df<-data.frame(x = 1:3, y = 3:1)
# 判断是否有叫x的列
> has_name(df,'x')
[1] TRUE
# 判断是否有行名
> has_rownames(df)
[1] FALSE
# 给df增加行名
> row.names(df)<-LETTERS[1:3];df
x y
A 1 3
B 2 2
C 3 1
# 判断是否有行名
> has_rownames(df)
[1] TRUE
# 去掉行名
> remove_rownames(df)
x y
1 1 3
2 2 2
3 3 1
# 把行名转换为单独的一列
> df2<-rownames_to_column(df, var = "rowname");df2
rowname x y
1 A 1 3
2 B 2 2
3 C 3 1
# 把一列设置为行名
> column_to_rownames(df2, var = "rowname")
x y
A 1 3
B 2 2
C 3 1
# 把行索引转换为单独的一列
> rowid_to_column(df, var = "rowid")
rowid x y
1 1 1 3
2 2 2 2
3 3 3 1
这些data.frame的工具函数,我猜是用于data.frame到tibble的数据类型转换用的,因为tiblle是没有行名的。
4. tibble的源代码分析
对于tibble包的深入理解,我们需要分析tibble包底层的源代码,以及设计原理。我们打开github上是tibble项目,找到tibble.R的源代码,先来了解一下tibble类型的定义。
找到tibble函数的定义:
tibble <- function(...) {
xs <- quos(..., .named = TRUE)
as_tibble(lst_quos(xs, expand = TRUE))
}
tibble函数的构成是非常简单地,用quos()和lst_quos()函数来分割参数,再用as_tibble()函数,生成tibble类型。
我们再找到as_tibble函数的定义:
as_tibble <- function(x, ...) {
UseMethod("as_tibble")
}
as_tibble.tbl_df <- function(x, ..., validate = FALSE) {
if (validate) return(NextMethod())
x
}
这个函数是一个S3类型的函数,可以S3面向对象类型的方法,来查找tibble相关的重写的函数。关于S3类型的详细介绍,请参与文章R语言基于S3的面向对象编程。
> methods(generic.function=as_tibble)
[1] as_tibble.data.frame* as_tibble.default* as_tibble.list* as_tibble.matrix*
[5] as_tibble.NULL* as_tibble.poly* as_tibble.table* as_tibble.tbl_df*
[9] as_tibble.ts*
利用S3的查询函数,把整个tibble类型定义的泛型化函数都找到了。
接下来,我们继续到tbl_df的类型的定义
#' @importFrom methods setOldClass
setOldClass(c("tbl_df", "tbl", "data.frame"))
最后,这样就明确了tbl_df是类的定义,包括了属性和方法,而tibble是实例化的对象。通过对tibble函数的源代码分析,了解tibble本身的结构是怎么样的。那么再接下来,就是如何利用tibble来进行用于数据科学的数据处理过程。请继续阅读下一篇文章:R语言数据科学数据处理包dplyr。
转载请注明出处:
http://blog.fens.me/r-tibble/